Desarrollo de Software
PLATAFORMA DE IMAGEN
- Inicio >
- Investigación >
- Plataformas tecnológicas >
- Imagen >
- Desarrollo de Software. Plataforma Imagen
- Servicio de soporte en Análisis y cuantificación de Imagen
- Estación de Trabajo de Análisis (Hab. B37)
- Estación de Trabajo de Análisis (Hab. B02)
Responsable:
Tomás Muñoz: tmsantoro@unav.es
Teléfono: +34 948 194 700 Ext. 1043
Análisis de imágenes biomédicas y médicas
Desarrollamos herramientas de software a medida de cada usuario, adaptadas para cada problema específico.
La mayoría de aplicaciones las desarrollamos como macros o plugins de Fiji/ImageJ. Se trata de una plataforma de software libre, lo que permite al usuario hacer uso de nuestras aplicaciones desde cualquier ordenador y realizar sus propios análisis y cuantificaciones de imagen.
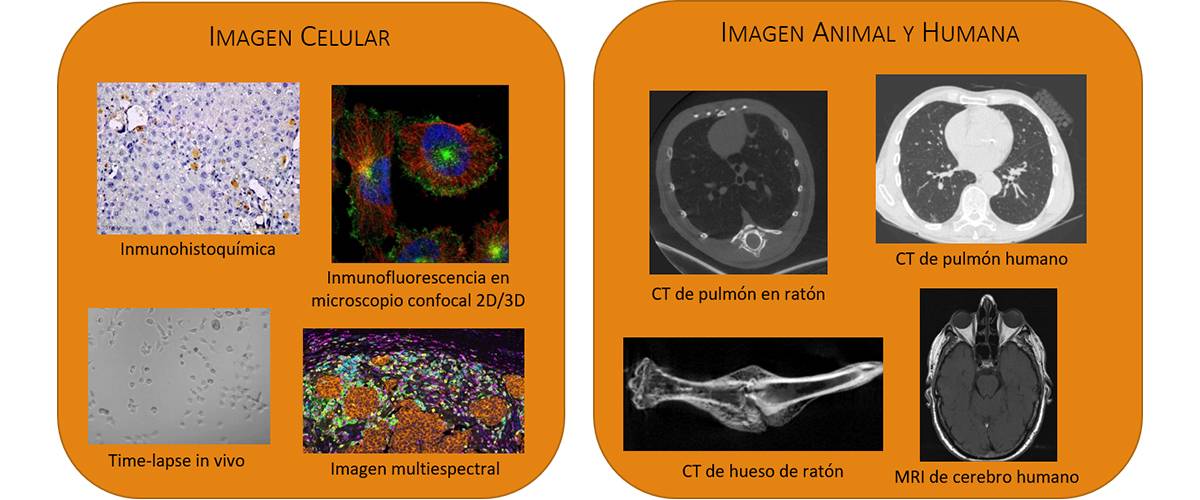
Trabajamos con diferentes modalidades de imagen

Extracción automática de cores individuales a partir de imágenes de TMA completo.
Los cores individuales son extraídos como imágenes individuales para un posterior procesado mediante otros plugins de Fiji.
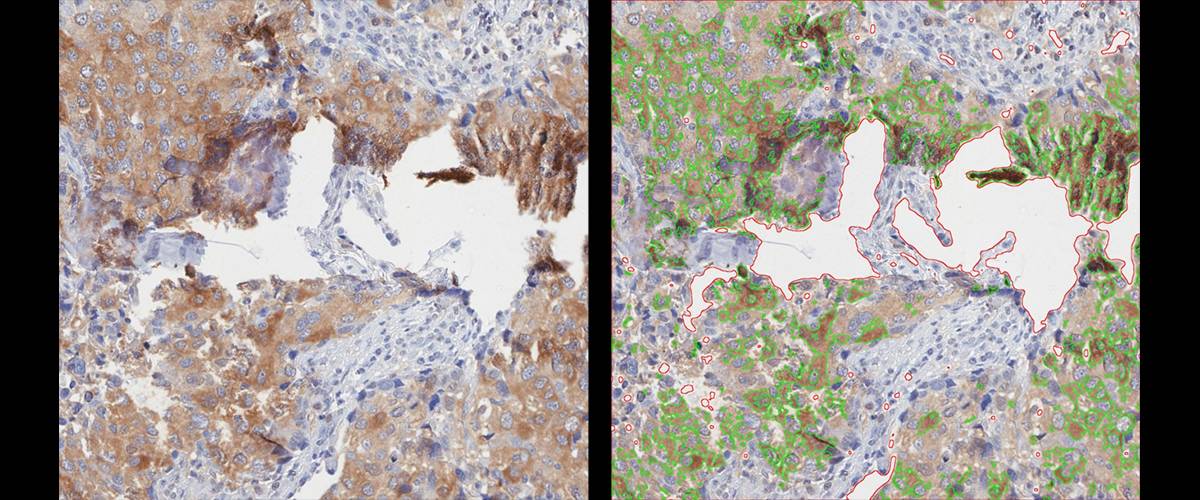
Detección y cuantificación de inmuno-tinción de DAB. Cuantificación de ratio de área positiva frente al área de tejido. Posibilidad de eliminación manual de regiones (zonas necróticas, pliegues, etc.)
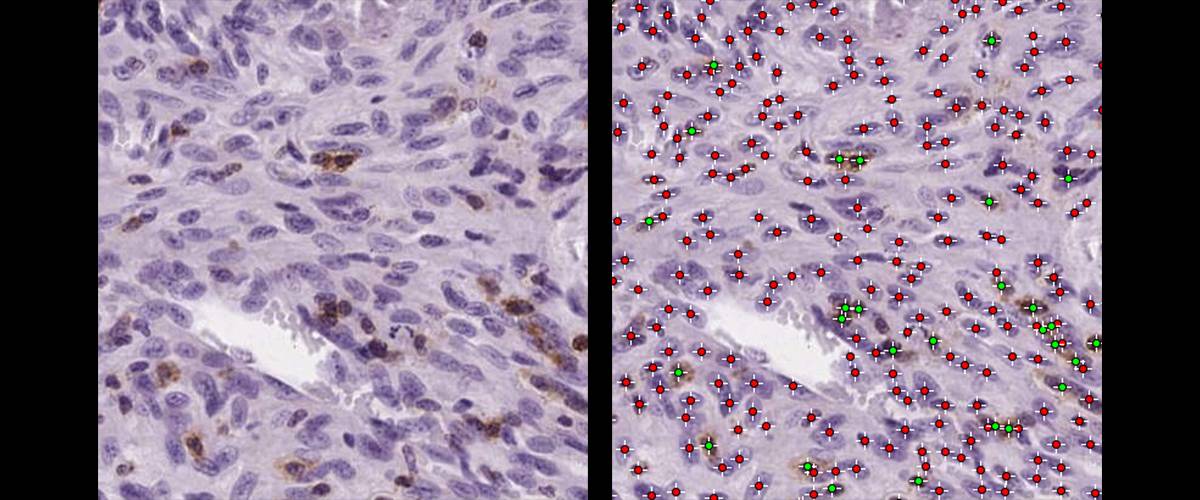
Detección y clasificación de células individuales en imágenes de H&E DAB. Clasificación y contaje de células positivas o negativas para DAB
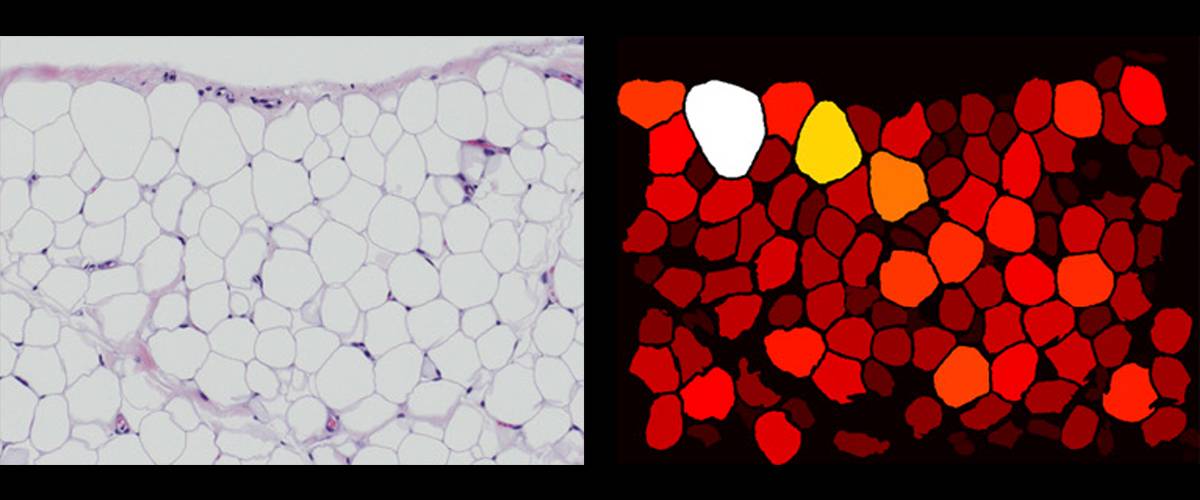
Adiposoft: software automatizado para el análisis de celularidad en tejido adiposo blanco en secciones histológicas. Segmentación de adipocitos individuales. Cuantificación morfológica de cada adipocito para obtener la distribución completa.
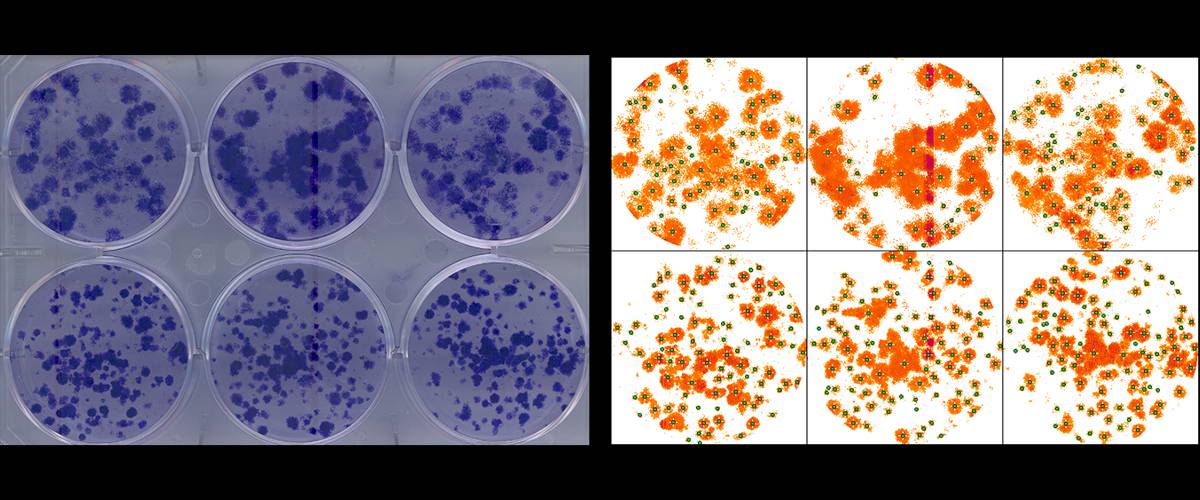
Análisis de ensayos de formación de colonias en placas de cultivo celular. Adaptación del plugin “ColonyArea” de Guzmán et al. 2014. Detección semi-automática de pocillos individuales, segmentación y cuantificación de área y número de colonias formadas en cada pocillo.
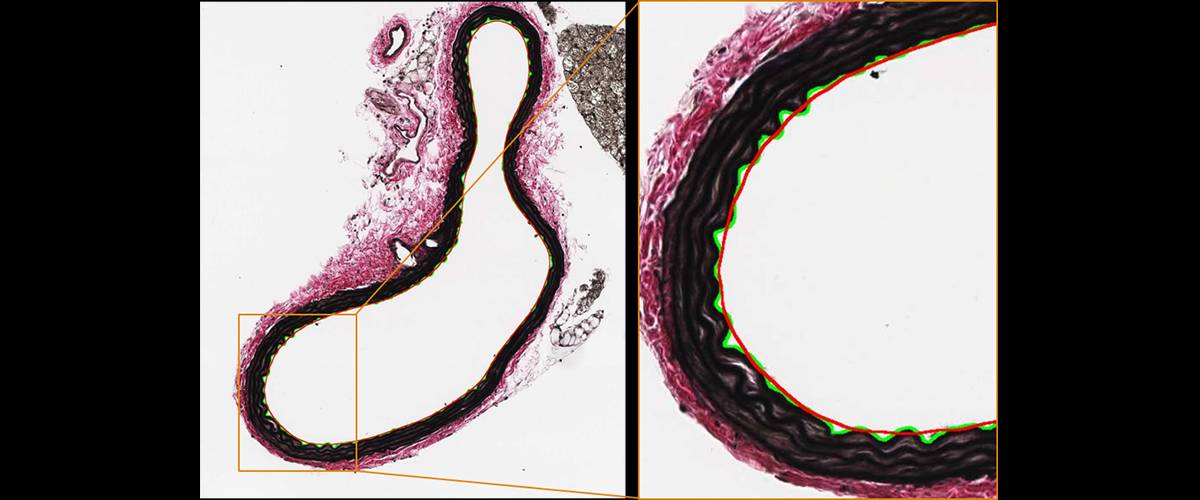
Análisis de la ondulación de la elastina en arterias. Detección automática del lumen de la arteria, de las ondulaciones de la elastina en el contorno interior, aproximación de un contorno sin ondulaciones y cálculo del ratio de longitud entre ambos contornos: ondulado (verde) vs. aproximación sin ondulaciones (rojo)
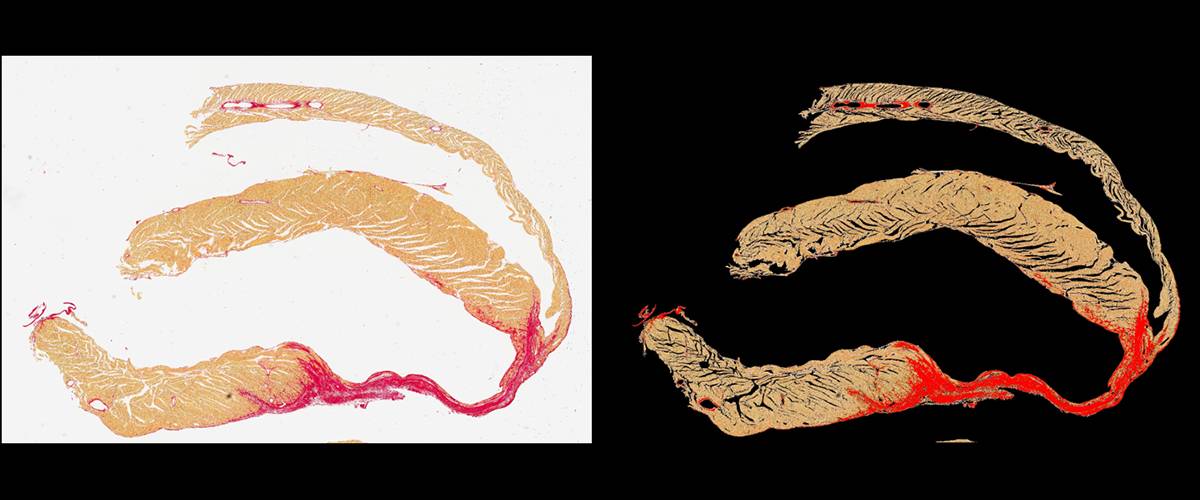
Cuantificación de tejido fibrótico mediante tinción de Rojo Sirio. Detección automática de tejido y de regiones con tinción positiva de rojo sirio. Cálculo de ratio de área positiva vs. área de tejido total.
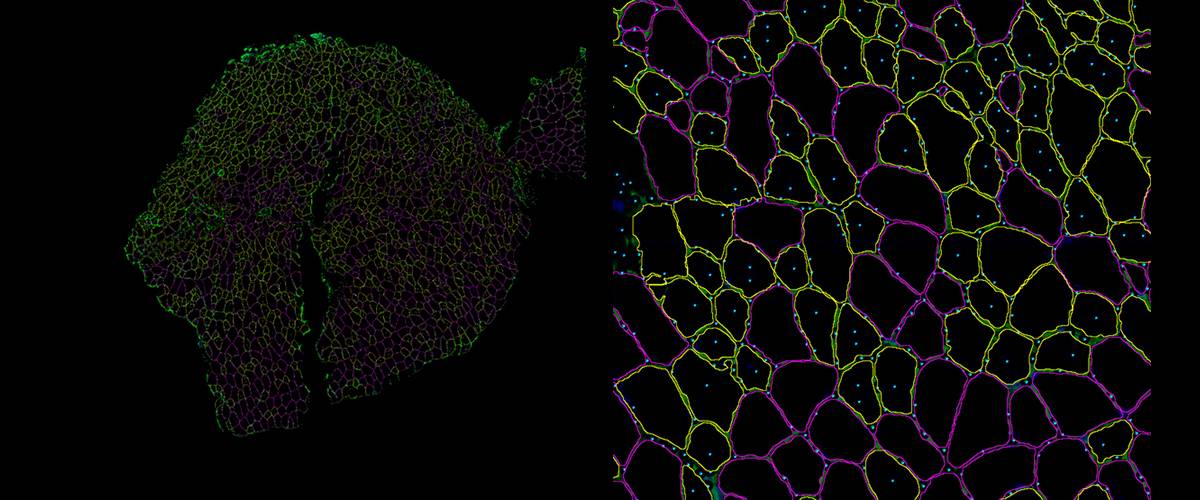
Detección, clasificación y cuantificación automática de fibras musculares en imágenes de inmunofluorescencia.
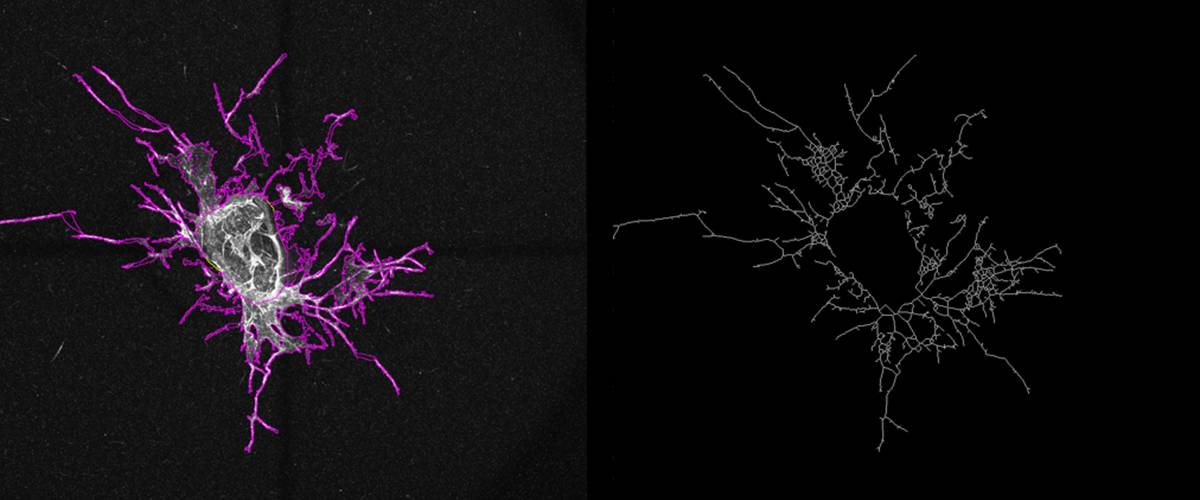
Cuantificación del crecimiento de vasos angiogénicos a partir de un segmento de la aorta.
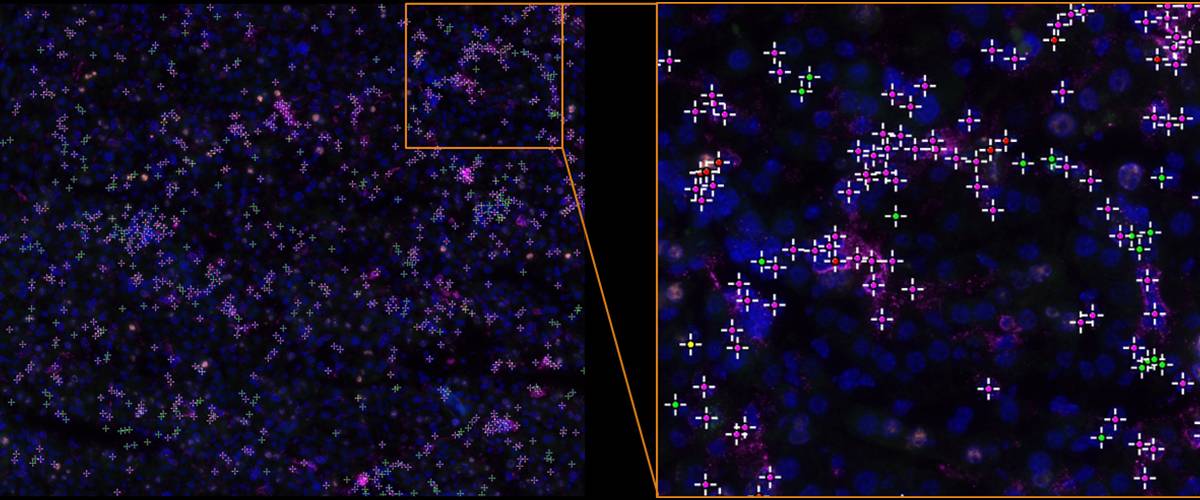
Cuantificación de poblaciones celulares en imágenes de fluorescencia multiplex.
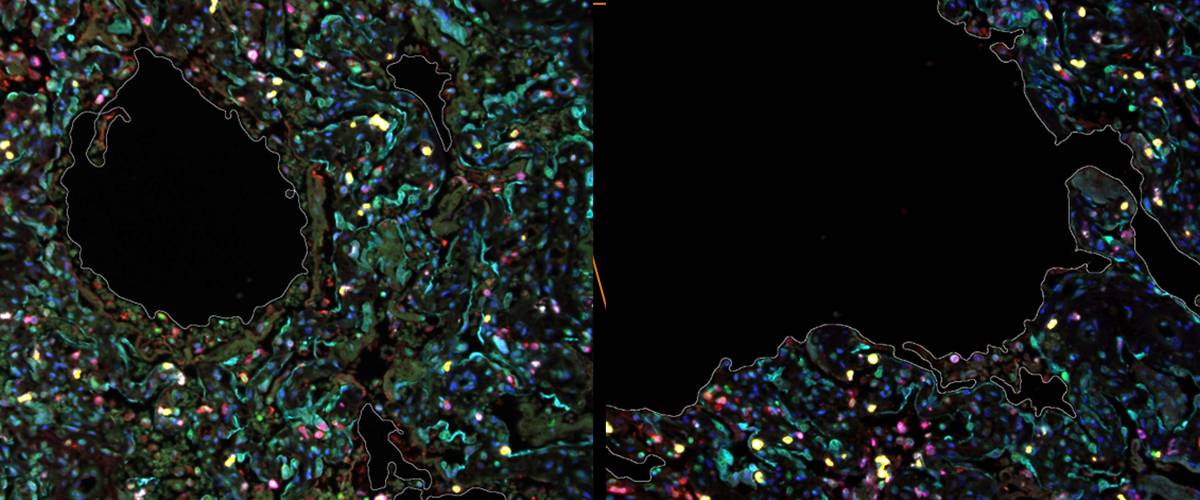
Detección de NETs en imágenes de fluorescencia multiplex mediante la localización de marcadores.
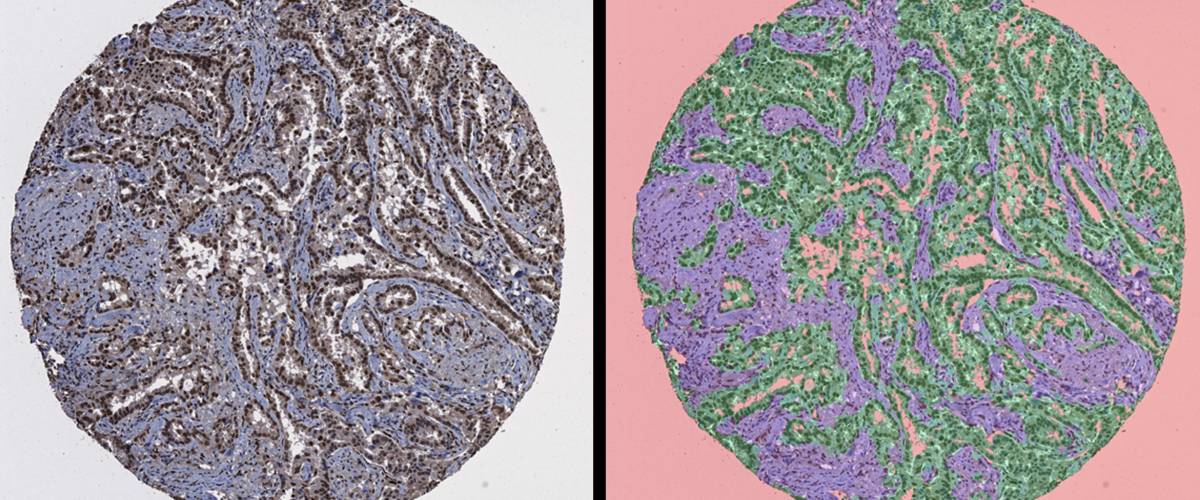
Segmentación automática de distintas regiones de tejido tumoral utilizando técnicas de machine learning.
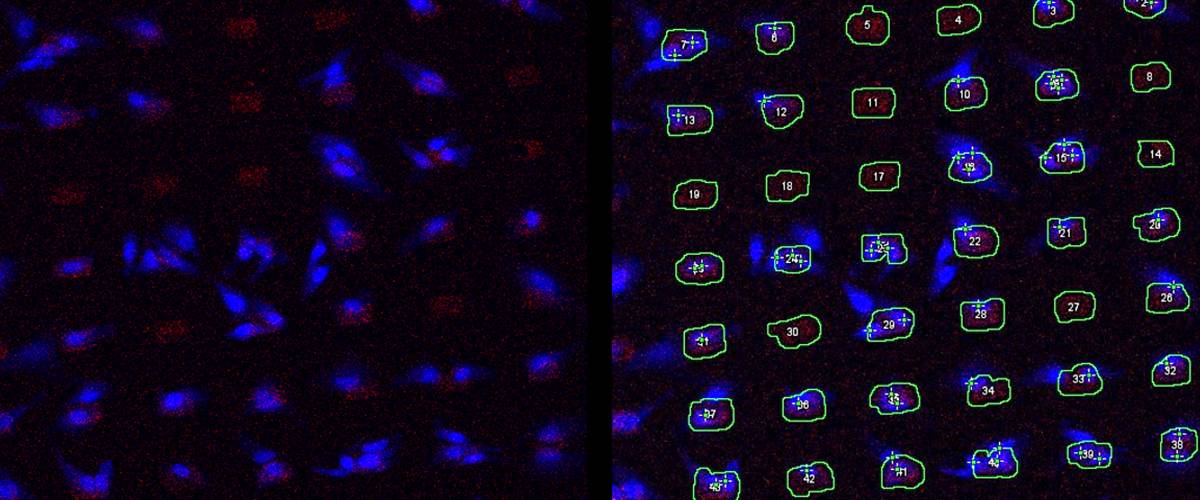
Detección de células tumorales sobre un patrón depositado por microcontact printing
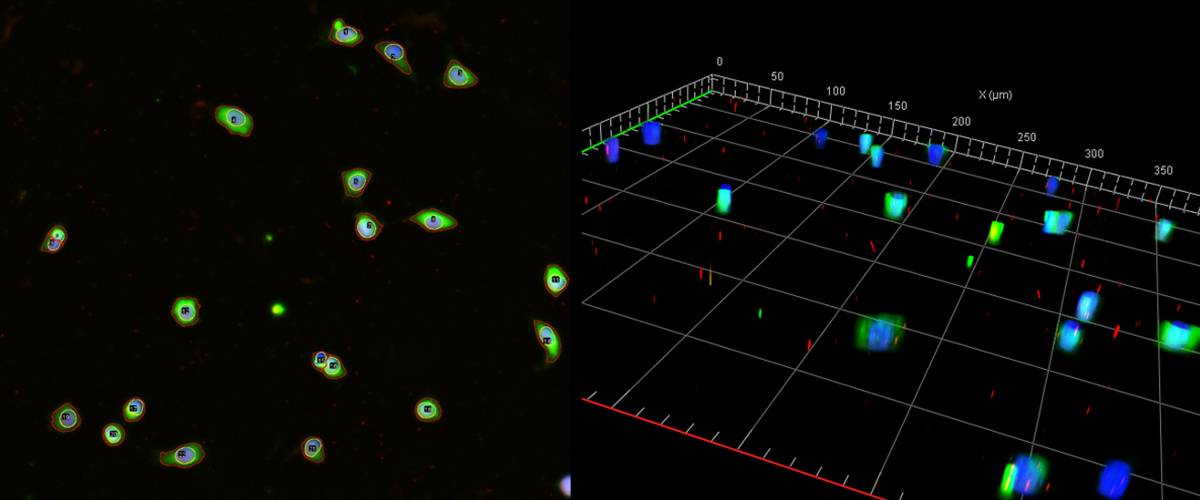
Cuantificación 3D de expresión de proteínas en núcleo y citoplasma en cultivos celulares.
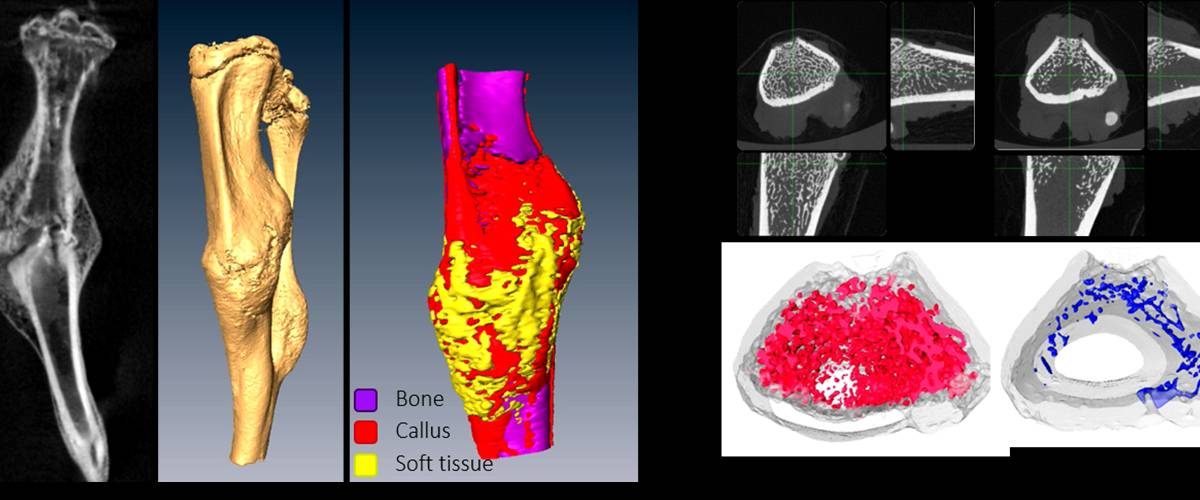
Segmentación y cuantificación 3D de estructuras óseas en imágenes de CT. Cuantificación de volumen y densidad ósea, y de parámetros histomorfológicos en el caso de las estructuras de hueso trabecular.

Segmentación y cuantificación 3D de pulmones y vías aéreas en imágenes de CT. Cuantificación de volumen e intensidad media pulmonar, con caracterización de grado de enfisema o de infección.
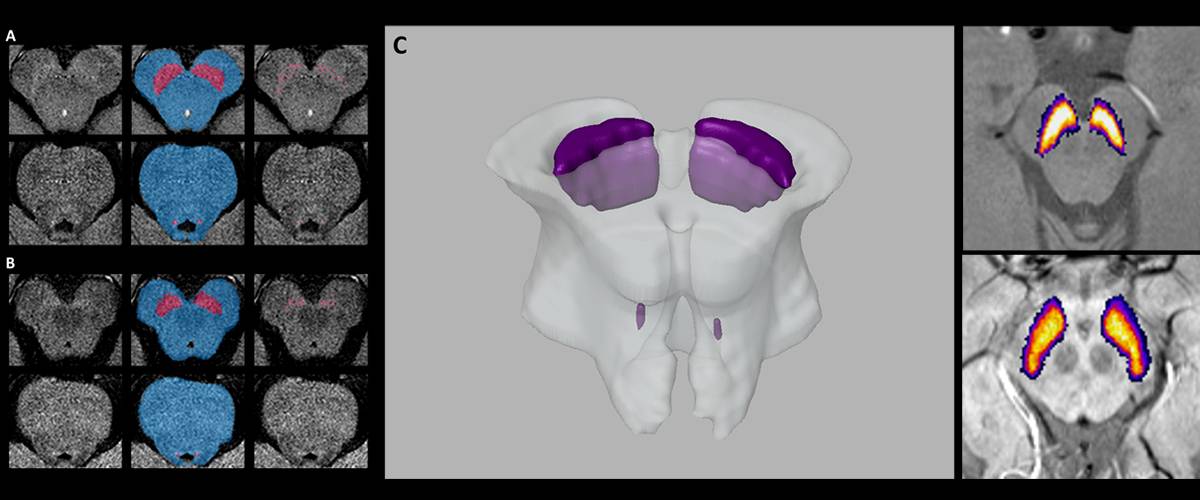
Segmentación y cuantificación de estructuras cerebrales en imágenes de MRI.
Segmentación y tracking 2D de células en dispositivos microfluídicos. Cálculo del track de cada célula a lo largo de la secuencia y cuantificación de parámetros de migración celular.
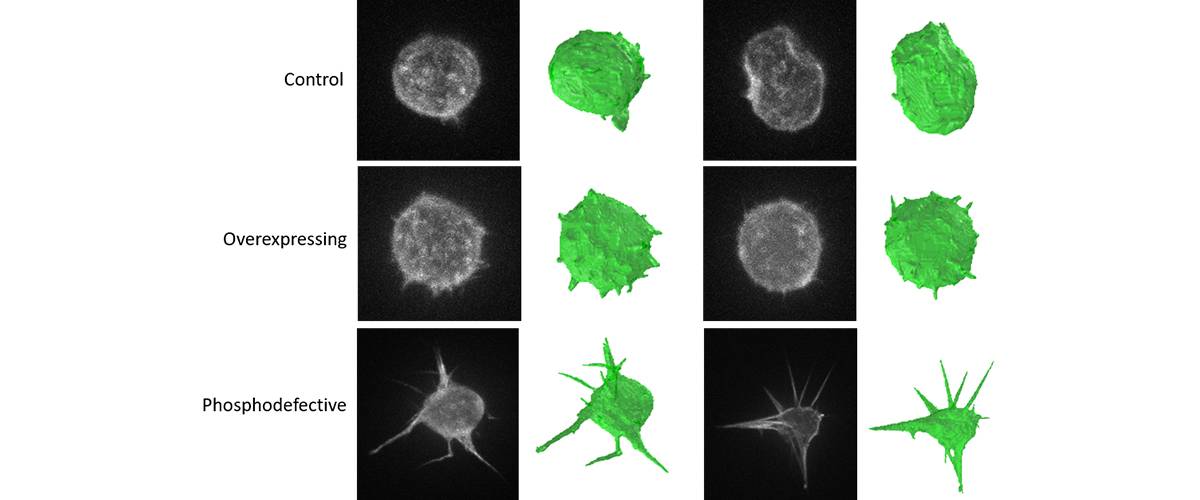
Segmentación y caracterización 3D de células de microscopía de alta resolución. Cuantificación morfológica y detección de filopodios en distintos fenotipos celulares.